MEGA4的中文使用说明
产生背景及简介
随着不同物种基因组测序的快速发展,产生了大量的DNA序列信息,这时就需要一种简便而快速的统计分析工具来对这些数据进行有效的分析,以提取其中包含的大量信息。MEGA就是基于这种需求开发的。MEGA软件的目的就是提供一个以进化的角度从DNA 和蛋白序列中提取有用的信息的工具,并且,此软件可以免费下载使用。
现在我们使用的是 MEGA4 的版本。它主要集中于进化分析获得的综合的序列信息。使用它我们可以编辑序列数据、序列比对、构建系统发育树、推测物种间的进化距离等。此软件的输出结果资源管理器允许用户浏览、编辑、打印输入所得到的结果而且所得到的结果具有不同形式的可视化效果。此外,该软件还能够得出不同序列间的距离矩阵,这是他不同与其他分析软件的地方。在计算矩阵方面有一些自己的特点:
1. 推测序列或者物种间的进化距离
2. 根据MCL(Maximum Composite Likeliood method)的方法构建系统发育树
3. 考虑到了不同碱基替换的不同的比率,考虑到了碱基转换和颠换的差别。
4. 随时可以使用标注:所以的结果输入都可以使用标注,而且标注的内容可以被保存,复制。
具体使用
我们以分析 20 个物种的血红蛋白为例来具体说明此软件的具体使用情况。
启动程序
1. 运行环境:在Windows 95/98, NT, ME, 2000, XP, vista 等操作系统下均可使用。
2. 下载安装:可以直接登陆 www.megasoftware.net 进行下载安装,另外还可以从http://abc.cbi.pku.edu.cn/tools/phylogeny.php 中的链接进去。
3. 双击桌面快捷方式图标, 进入主界面;或者从开始菜单,单击图标启动。
序列分析
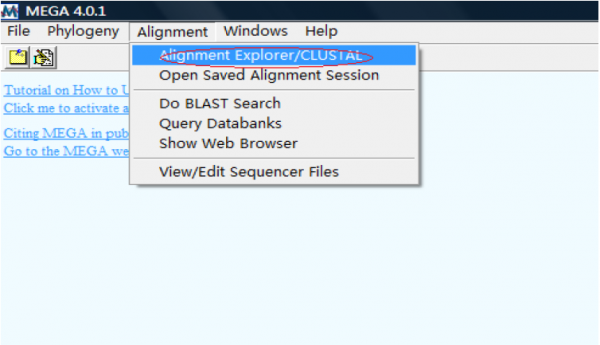
1.启动
单击后,会出现如下界面:
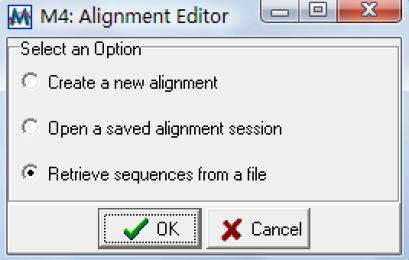
这里有三个选项,分别对应三种不同的情况:以下分别予以介绍:
Create a new alignment :是在你没有任何比对的时候使用,比如你只有一个fasta 格式的序列就可以选择这个选项。
Open a saved alignment session:使用它可以打开一个我们已经比对好的序列文件;
Retieve a sequence from a file :这种情况同第一种情况相似,只是不用选择是DNA 还是蛋白质序列比对,选择的也是fasta 格式的文件,打开后的界面都是一样的。
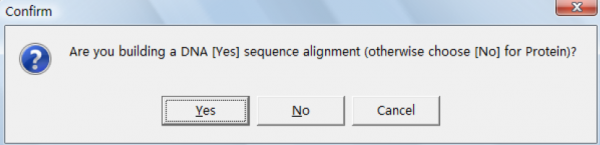
以第一种情况为例说明,点击如出现下界面:
这里我们分析的是蛋白序列所以选择 No。然后从 data 菜单选择输入数据文件如图:
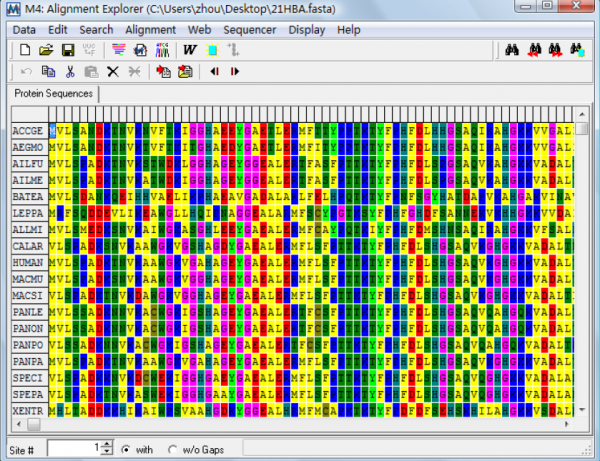
选择你保存的 fasta 格式序列后就会出现:
菜单的使用
Data菜单
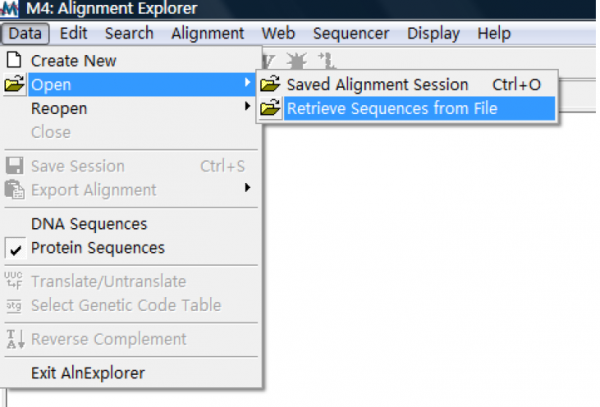
Creat a new :创建一个新的数据比对文件,也就是说当我们比对完一组后,想接着比对另一组,那么使用它就可以不用退出直接把数据文件导入;
Open:打开先前已经比对并保存好的文件,它包含两个子菜单:retive sequence from file 和saved aligment session ;
Close: 关闭当前的比对数据文件;Save session:保存当前比对结果,可以给比对的结果一个文件名;
Export alignment:将当前的序列比对结果输出到指定文件,有两种输入格式可供选择:MGTA 和FASTA.
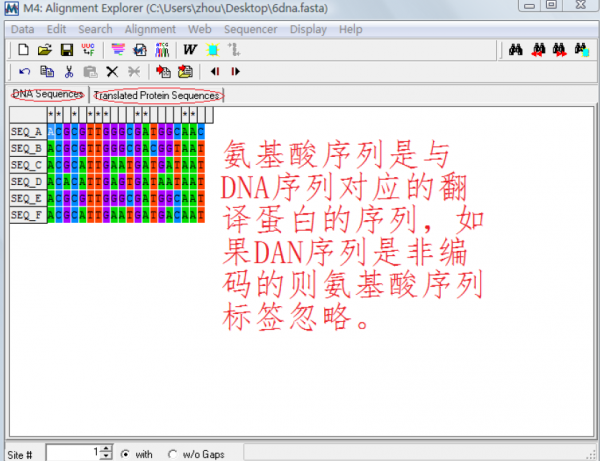
DNA sequence:使用它来选择输入的数据DNA 序列,这里需要说明的是如果你输入的数据是氨基酸序列的话,比对窗口只显示一个标签,若是DNA 序列的话则显示两个标签,一个是DNA 序列的,另一个是氨基酸序列的。如下图:
Protein sequences:选择输入的氨基酸序列,选择后,所以的位点就被当作氨基酸残基位点来对待。
Translate/untranslate:只有比对的序列是编码蛋白的DNA序列的时候才可用。它可以根据指定的遗传密码表将DNA 序列翻译成特定的氨基酸序列。
Select genetic code table:使用它将编码蛋白的DNA 翻译成特定的蛋白序列。
Reverse complement:将选择的一整行的DNA 序列变为与之互补配对碱基序列。
Exit alignment explorer:退出序列比对的资源管理窗口。
Edit 菜单
使用这个菜单可以对我们的比对序列进行想要的一些编辑工作具体为
Undo:撤销上一步操作;
Copy:复制;cut:剪切;Paste:粘贴;前面三个操作都可以只针对一个碱
基或氨基酸残基也可以是一段甚至是整个序列;
Delete:从比对表格中删除一段序列;
Delete gaps:去掉序列中的空缺;
Insert blank sequence:重新插入一空行;标签和序列都是空的;
Insert sequence from file:从已保存的文件中插入新的序列;
Select sites:选择一列序列,与点击比对表上方的灰白空格作用类似;
Select sequence:选择一行序列,与点击比对表格左侧的标签名作用类似;
Select all:全选;
Allow base editing:只读保护,只有选择后才能对序列进行编辑操作,否则所以的序列为只读格式,不能进行任何编辑操作。
Search 菜单
用来快捷查找序列中的标记未定或者目的碱基或残基。
Find motif:选择后出现如下对话框:
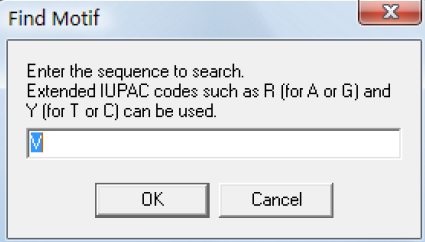
输入你想要查看的一小段序列。找到后会以黄色标出;
Find next:在序列的下游查找目的序列片段;
Find preious:在序列的上有查找目的序列片段;
Find marked sites:查找标记位点;
Highlight motif:突出标记已经选择的位点。
Web 菜单
这个菜单提供一个链接 Genbank 的入口,可以在网上直接做Blast 搜索。当手上没有准备好要比对的序列时,可以直接去网上搜索。
Query gene banks:开启NCBI 的主页;
Do blast search:开启NCBI BLAST 主页;
Show browser:开启网页浏览器。
Sequencer 菜单
此菜单下只有一个子菜单:edit sequencer file,用来打开一个打开文件对话框,此对话框可以打开一个sequencer data file,一旦打开,这个文件就在trace data file viewer/editor 的对话框中展示出来。这个编辑窗口允许你查看和编辑automatd DNA sequencer 产生的trace data。它可以阅读和编辑ABI 和Staden 格式文件并且序列可以直接被导入到序列比对窗口或被上传到网页浏览器做blast 搜索。
Display 菜单:
这个菜单相对简单,主要用来调整工具栏。
Toolbars:工具栏菜单,它包含一些子菜单,选择后就会出现在比对的窗口中;
Use colors:将不同的位点以不同的颜色显示;
Background color:选择后位点的显示与位点一样的背景颜色;
Font:字体对话框,通过选择来调整窗口中的序列字符的大小。
实例介绍alignment 菜单
Mark/unmark site:在比对的表格中标记或者不标记一个单一位点,一次每条序列只能被标记一个位点,不同序列间的位点你可以选择同一列的,也可以是错开的,要根据自己的目的进行选择。选择标记后的序列可以使用align marked sites进行比对分析。
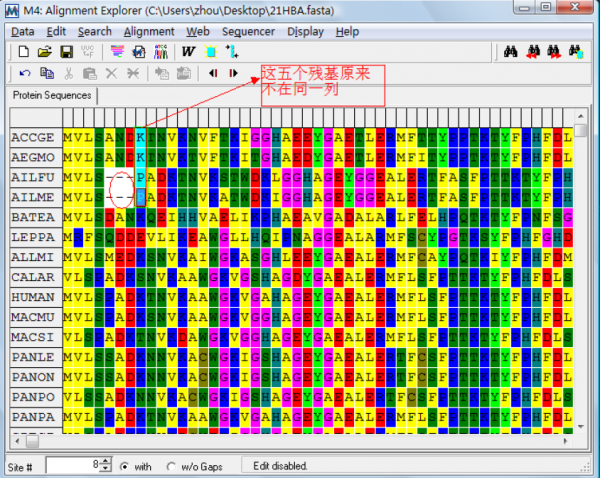
Align marked sites:比对标记的序列,在这里如果在两个或多个序列间标记了不在一列的位点重新比对后会出现空格。如图:
Unmarked all sites:把所以标记的位点去标记;
Delete gap-only site:去掉序同是空格的一列;这在多序列比对前很有用。
Auto-fill gaps:使用空格补齐不同长度的序列。
Align by ClustalW:此软件整合了clustalw 程序,这也是它的方便之处,选择
要比对的序列后点击会出现下面的对话框:
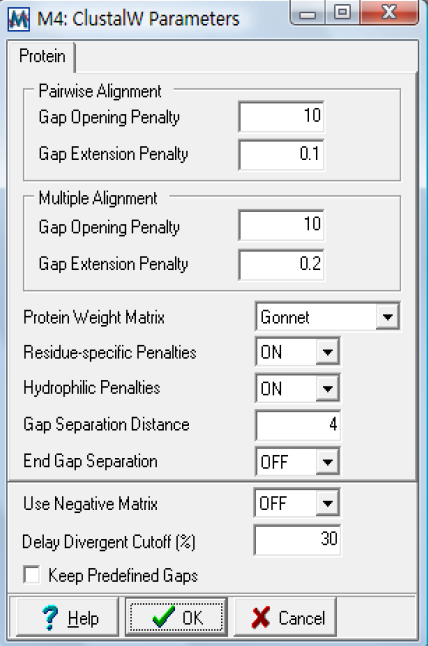
这是一个序列比对参数设置对话框,需要注意的是:这个软件不会考虑到核酸序列中的编码位点,所以在比对的过程中可能会在编码区中插入空格,所以如果分析cDNA 或者编码序列建议将他们翻译成蛋白序列后在比对。
一对序列比对和多序列比对下的设置都是一样的如下:
Gap opening penalty:空格罚分设置,增加一个空格就罚相应的分值,增加这一分值会降低空格出现的频率。
Gap extension penalty:空格扩展罚分,就是根据空格的长度来罚分,增加这一分值会使空格变短,末端空格不计入罚分。
一般参数:
DNA/protein weight matrix:选择不同的加权矩阵;
Residue-specific penalties:特殊氨基酸罚分。在序列比对的过程中特异氨基酸可能增加或减少罚分值,比如:富含甘氨酸的区段比富含缬氨酸的区段更可能有空格出现,因而他们的罚分不同。
Hydrophilic penalties:如果有连续的5 个或者更多的亲水性氨基酸的话,他们倾向于出现空格,这些区段很可能出现环状或卷曲,因此罚分不一样。
Gap separation distance:参数设置来尽可能降低空格之间离的太近的机会,小于指定数值的空格罚分要多余其他的,这不能避免出现相邻空格,只能降低他们出现的频率。
Use negative matrix:使用负性矩阵,
Delay divergent cutoff:若一条序列相似性低于设定的百分值将推迟比对。
当一切参数都设定好了之后就点击 OK 就可以进行比对了,中间出现一个过度对话框。比对结束后,可以将结果保存(data/save session/),以供构建系统发育树使用。另外,如果不保存直接关闭,系统跳出一个确认对话框。
下面这个是序列数据管理的管理界面,此外我们还可以通过主界面上的data/open data 路径打开,效果是一样的,注意这里打开的只能是刚才保存的后缀是.MEG 的文件。
当这个序列数据界面出来后,注意软件的主界面发生了一定的变化,多出了几个功能菜单:
下面就着重介绍一下序列数据窗口的一些具体使用:
这个窗口用来展示比对后的序列数据,这里提供了许多的功能菜单用来查看序列比对后的数据统计结果或者来选择想要的子序列。
Data 菜单
Write data to file:导入序列打开窗口;
Translate/untranslate:将蛋白编码序列翻译成蛋白序列,或者再转变成核酸序列;
Selected genetic code table:打开select genetic code 对话框,从这个对话框可以选择编辑或者添加遗传密码表;
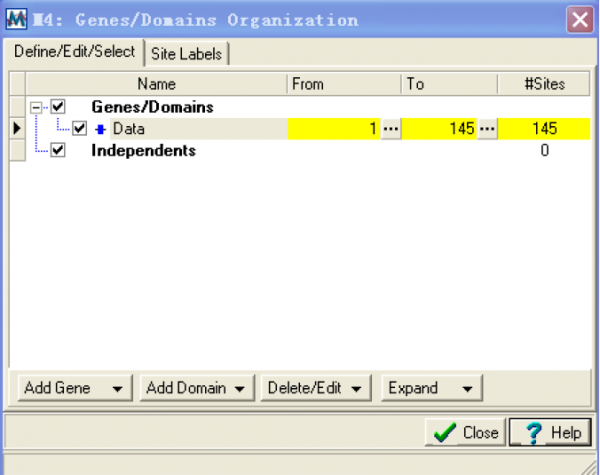
Setup/select genes and domains:打开sequence data organizer 对话框,在这个对话框里可以定义和编辑基因和结构域。
使用这个窗口可以查看,定义,和选择结构域和基因,并且标记单个的位点。具体使用这里不作详细介绍。
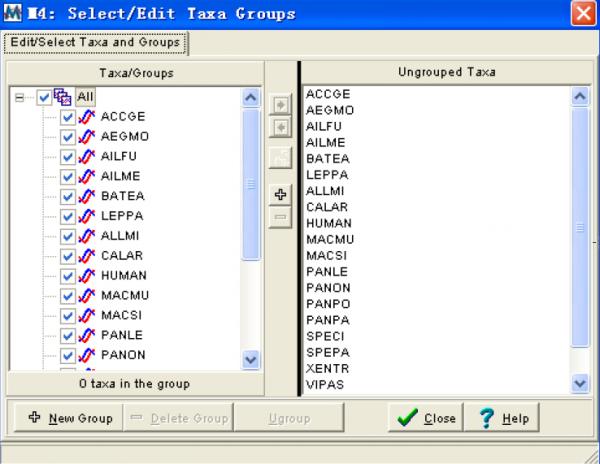
Setup/select taxa and groups:打开一个可以编辑分类和定义分类组的对话框:
这个窗口分为两个子窗口,左边的是分类组,显示不同的分组情况,右边的是未分组窗口显示还没有归入任何一个组群的分类。中间和下边是一些操作键,通过他们我们可以建立新的组,如果你将所以的分类都归入到不同的组里,并且给予组名,你们在序列数据窗口中就会在物种名字后边显示他所属的组名。
Display 菜单
Show only selected sequence:只显示你所选择的感兴趣的序列;
Use identical symbol:将一列中绝大部分相同等碱基或氨基酸字符用点来代替;
Color cells:将序列中连续的一致的碱基或者氨基酸给以相同的颜色背景以区别显示;
Sort sequences:将显示的分类以不同的方式排序,可以根据序列名字、组名来排序;
Restore input order:将经过修改的序列顺序回复到刚打开时的样子;
Show sequence name:显示序列的名字,不选则隐藏;
Grouped:显示组名;
Change font:更改显示的字体格式。
Quite data viewer:退出界面。
Highlight 菜单
这里的子菜单大部分都显示在工具栏里,如图所示:
分别是高亮度显示保守序列、可变序列、比对信息序列、和一列中至少有两个不同字符的列等。
Statistics 菜单
Nucleotide composition:当序列为核酸时可用。计算每条序列中的不同的碱基百分比;
Nucleotide pair frenquencies:只有当序列为核酸时可用。
Codon usage:只有序列为编码蛋白的核酸序列时可用。计算出codon usage的百分比和RSCU(relative synonymous codon usage)值;
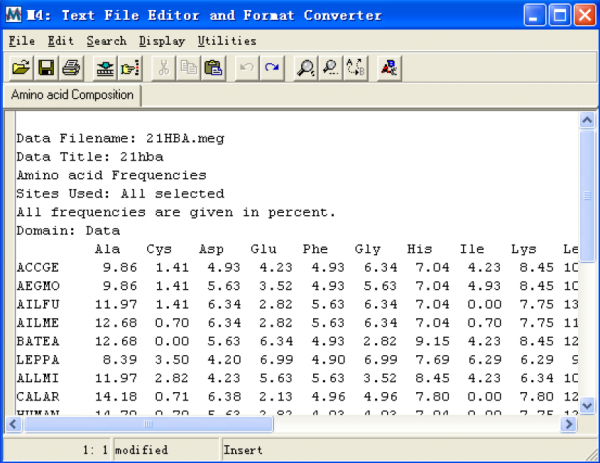
Amino acid composition:当序列为氨基酸序列或编码蛋白的核酸序列时可用。计算每条序列氨基酸残基的百分比;并且跳出一个显示窗口,在这个窗口中可以进行许多操作:可以得到的这一数据保存到文件中;还可以打印出来;还以直接分析统计所
得到的结果,查看每一行等。具体大家可以自己摸索;
Use all selected sites:保证上面的分析统计是在选择所有的序列下进行的,不考虑被标记的位点。
从以上大家应该可以粗略的了解到这个软件的强大而又方便的序列比对分析的功能。下面再简要介绍主页面上的几个菜单的使用。
Distances 菜单
相关原理:两条序列间的进化距离是通过计算两条序列间碱基或氨基酸替换得来的,推测进化距离是研究分子进化、构建系统发育树和推测物种分化时间的基础,这个软件中包括了绝大部分广泛使用的推测进化距离的方法。值得提出的是,该软件还使用解析公式和bootstrap 的方法来评价出现的错误。
该软件所包括的方法大致可被分为三类:核酸;同义—非同义替换;氨基酸。
1) 核酸:序列是核酸和核酸之间的比较,计算编码蛋白和非编码蛋白的核酸序列间的进化距离,主要有两种方法: No. of differences 和p-distance 还包括许多的模型:Jukes-Cantor Model 、Tajima-Nei Model、Kimura 2-Parameter Model、Tamura 3-Parameter Model、Tamura-Nei Model、Maximum Composite Likelihood Model 等,可以根据需要进行不同的选择。
2) 同义-非同义替换:序列是编码子和编码子之间的比较,所以只能用来计算编码蛋白的序列。常用的模型有: Nei-Gojobori Method 、 Modified Nei-Gojobori Method 、Li-Wu-Luo Method 、 Pamilo-Bianchi-Li Method、Kumar Method 等。
3) 氨基酸类:序列间是氨基酸残基之间的比较。能够用来计算氨基酸序列间以及编码蛋白的核酸间的距离,编码蛋白的核酸在比对的时候自动被翻译成氨基酸序列进行比较。常用的模型有:Poisson Model、Equal Input Model、 Dayhoff and JTT Models。
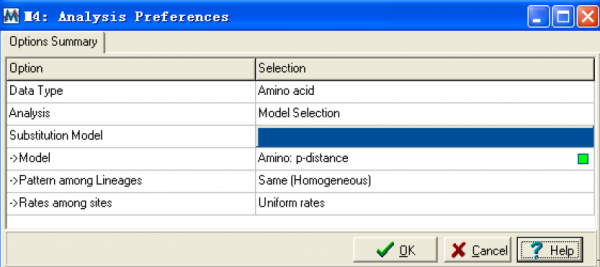
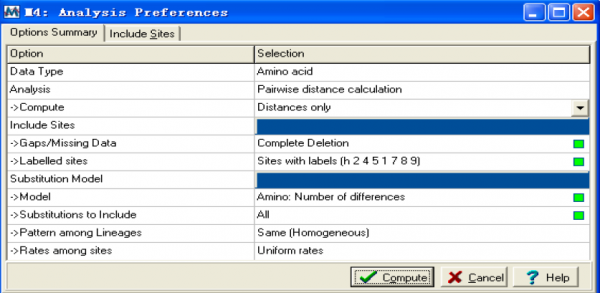
Choose model…:选择模型,选择跳出一个距离模型的选项窗口:
在这个窗口里,model 选项是选择推测进化距离的随机模型的,可以通过单击绿色小方框进行选择。Pattern among lineages:只有当距离模型选定后才可用;
rates among sites:允许位点间存在不同的替换率。选好后单击OK 即可。
Compute pairwise:单击出现上面类似的对话框:
Compute:选择是只计算进化距离还是选择计算同时进行评价。选择后者会出现standard error computation by 选项,通过这一选项选择解析公式或者bootstrap method 来评价结果的好坏。Gaps and missing data:在计算开始前选择去除所有包含比对空格和失意的位点;
另外,最初你也可以保留这些位点,在必要的时候在去掉。Labled sites:只有当一些或者全部位点有相关标签时才可用。
点击绿色方框,就可以看到包括选择标签的位点,如果你选择这些位点的话,这些位点就最先从数据中提出来。选好后compute 出现以下窗口:
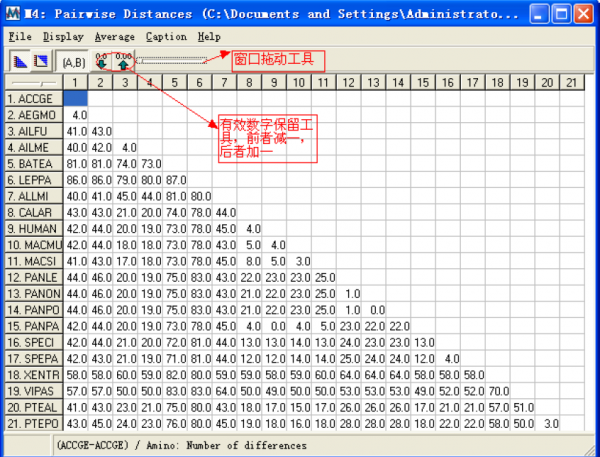
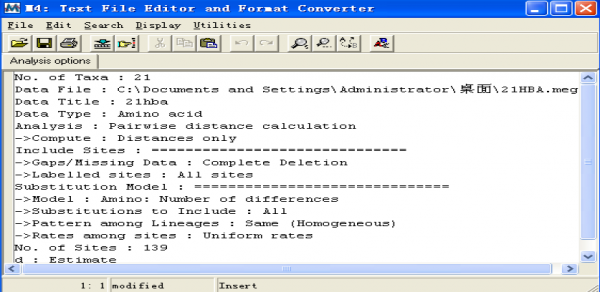
这是一个比对后的距离矩阵窗口,这个窗口包括很多不同的功能菜单,来调节显示的内容。File 菜单中有一个子菜单是Show Analysis Description:显示计算所用的不同的选项,这些信息可以被保存或者打印出来。
Average Menu:这里面有个子菜单Overall 单击会显示比对的总体平均距离。
Distance 菜单中其他的子菜单操作同上类似只是内容略有不同,具体可自行摸索。
Phylogeny菜单
Phylogeny 选项中有以下子菜单:

其中 Construct Phylogeny 和Bootstrap Test of Phylogeny 基本一致,其中后者给出了在计算过程中的出现的概率。
最大简约法 Maximum Parsimony,使用的运算法则是branch-and bound 的检索方法。得到的是无根树。这种方法在序列非常相似以及序列数目较小的情形下较适用(构建21条序列的进化树时,在几种方法中花费的时间最长)。
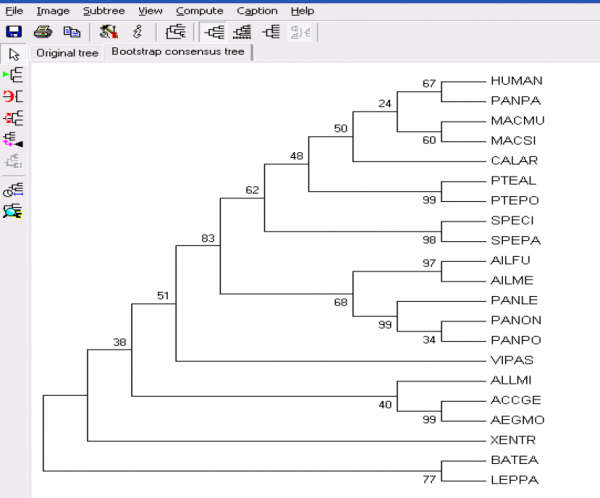
在实际运行得到拓扑图之后,上面有两个选项,点击 Original tree,可以选择查看计算所得到的所有结构树。
点击 Bootstrap consensus tree 得到我们所需要的结果

邻接法 Neighbor Joining: 当所考虑的谱系间进化速率可变时,邻接法特别适用。邻接法能给出枝长最小平方估计的序列,即能最真实的反映序列间的真实距离。邻接法得到的进化树也是无根树。邻接法有6 种计算方法,分别是No. of Differences、p-distance、Poisson Correction、Equal Input、PAM Matrix (Dayhoff)、JTT Matrix (Jones-Taylor-Thornton)。通常选
择p-distance。
最小进化法 Minimum Evolution:该方法和邻接法基本相似,在此不作介绍。
算术平均的非加权对群法 UPGMA:它假设沿着进化树分支的变化速率为一个常数,而距离近似为非加权的。UPGMA 法由计算关系最近序列间的枝长开始,然后计算序列对与下一个序列对间的距离平均值,不断重复直到所有序列都被包括在树中。如果树枝间的突变率不一致时,UPGMA 法将导致一个错误的树,因此该法现在已基本不用。
Relative Rate Tests
点击 Tajima’s Test,得到下面的对话框。
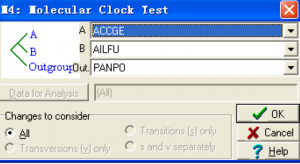
我们可以在对话框中选择比对序列中的任意三条序列,点击OK 之后,可以得到这三条序列进行比对的一些基本信息。
因此当我们得出系统发育树时,如果对其中的一些分支存在疑问,就可以将该分支序列进行Tajima 检测,帮助我们得出正确的结论。