ORF Finder使用说明及基因预测
2014-07-22
ORF Finder是一个图形的序列分析工具,分析并找到序列的ORF区(开放读码框架),这个工具使用标准的或其它特殊的遗传密码子列出所有可能的ORF区,并推导出氨基酸序列。
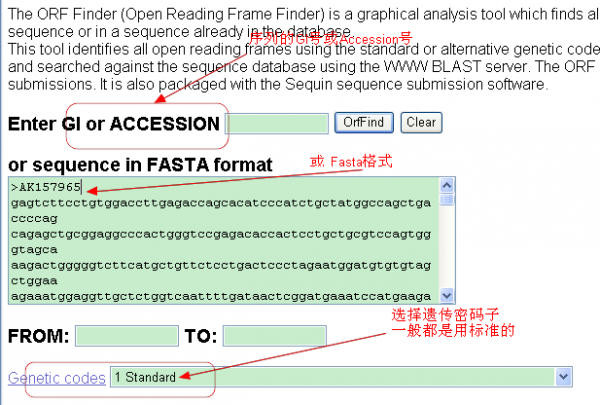
在线网址:http://www.ncbi.nlm.nih.gov/gorf/gorf.html
1,输入GI号或Accession,或直接输入序列的fasta格式
2,结果出现六个图形,这是根据六种不同的编码方式得到的(包括正反链)。右边出现各个预测的ORF区的长度与编码方式。点其中一个就可以该区域的序列。并且有推导的氨基酸序列。一般来讲,长的ORF区基本上都是正确的,有可能编码基因。下面介绍也可以根据其它工具来验证。
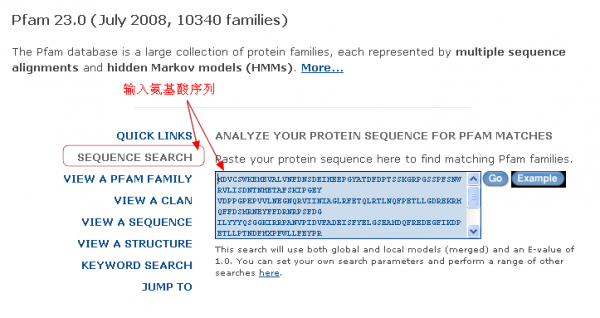
3,拿到氨基酸序列后,你可以直接做blastp,如果有匹配到,就是正确的ORF区了。另外也可以用Pfam的方法,在Pfam数据库搜索。
如:http://pfam.sanger.ac.uk/ ,输入氨基酸序列,仅仅是序列就可以了。匹配到的话就会有结果,说明是属于那个家族的,能匹配就说明是正确的。
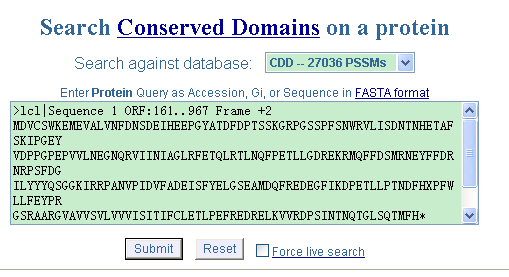
4,另一个工具是NCBI的CCD工个,网址:http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi ,同样输入氨基酸序列。
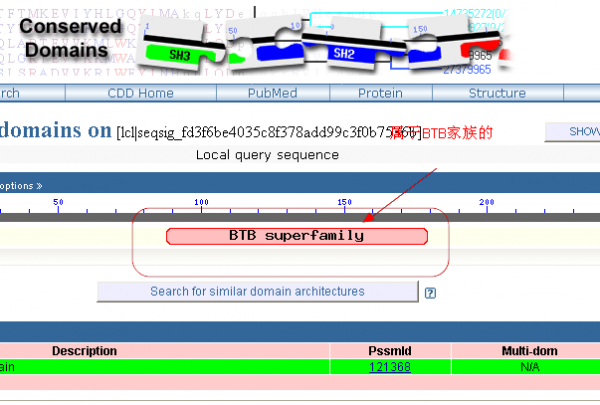
5,结果表明该ORF编码的蛋白是属于BTB家族的。
说明,你可以同时用几个工具来验证,如blastp,Pfam,CDD。但有时的结果不一定全部一致。这有可能是因为不能的数据库,存放的数据有些差异,算法也有些差异,是属于正常的。基本上大部分都是可靠的。